型号:内详
联系人:李先生
联系电话:18618101725
品牌:法国 以及美国flexercell
销售欧美进口各种不同基底静态培养及不同基底力学刺激环境动态培养装置
一、法国基底刚度可调控微图案培养产品
特点:
控制细胞的3D结构和力学
细胞在平坦或微结构化的软3D环境中培养,以模仿体内条件。
基材的刚度可以从非常软(1 kPa)到非常硬(200 kPa)中选择
提供多种基材形貌(平坦,圆形孔,方形孔,凹槽等)
基于凝胶的底物已准备好用于您的细胞培养实验
由于细胞直接接种在特征的顶部(易于限制非迁移细胞),因此易于使用且易于使用
预涂ECM基质(例如纤连蛋白)
适用于任何细胞培养底物(盖玻片,培养皿,多孔板)
凝胶的光学透明性使这些底物与高分辨率光学显微镜系统兼容

-
Cardiac spheroids as promising in vitro models to study the human heart microenvironment
Polonchuk, L., et al. Scientific reports, 2017 7(1), 7005 -
Current and emerging modalities for detection of cardiotoxicity in cardio-oncology
Khouri, M. G., et al. Future cardiology, 2015 11(4), 471-484 -
Bioinspired living structural color hydrogels
Fu, F., et al. Science Robotics, 2018 3(16), eaar8580
-
Preparation of hydrogel substrates with tunable mechanical properties
Tse, J. R., & Engler, A. J. Current protocols in cell biology, 2010 47(1) 10-16 -
Differentiation of liver progenitor cell line to functional organotypic cultures in 3D nanofibrillar cellulose and hyaluronan-gelatin hydrogels
Malinen, M. M., et al. Biomaterials, 2014 35(19), 5110-5121.
可订购的产品:
1、涂有凝胶盖玻片COVERSLIPS
---调整基材的刚度以重现体内环境
提供用于细胞培养的聚丙烯酰胺凝胶涂层盖玻片。
可用的基材刚度范围类似于细胞自然嵌入的体内机械性能。
由于盖玻片已准备就绪,可以轻松快速地培养细胞。
多种刚性:
从非常软(1 kPa)到非常硬(200 kPa)
随时可用:
直接接种细胞
光学透明:
易于光学观察
涂有纤连蛋白
与高分辨率光学显微镜系统兼容
在单的塑料袋中以水基溶液形式提供,以保持凝胶的特性
标准尺寸:24 mm圆形盖玻片(约170μm厚度)
与灌注室兼容
规格需求可定制;
涂有凝胶盖玻片COVERSLIPS目录:

涂有微结构凝胶的盖玻片
---同时控制形状和刚度以重现体内环境

提供用于细胞培养的微结构聚丙烯酰胺凝胶涂层盖玻片。
基于凝胶的基质包含呈开放微通道(凹槽)或孔形式的软或刚性微结构。
因此,可以在模拟体内环境的地形特征和刚度的底物上培养细胞。
>随时可用
直接接种细胞
>多种刚性
从非常软(1 kPa)到非常硬(200 kPa)
>广泛的3D设计
沟槽,方孔,圆孔等
涂有纤连蛋白,因此可以使用
与高分辨率光学显微镜系统兼容
在单的塑料袋中以水基溶液形式提供,以保持凝胶的特性
提供标准形状:凹槽和圆孔
标准尺寸:24 mm圆形盖玻片(约170μm厚度)
与灌注室兼容


标准的24毫米圆弧形凹槽和凹槽的典型布置:
每个盖玻片上有八个区域,其特征是宽度(凹槽)或直径(圆孔)不同:从10μm到100μm。
结构的深度为10μm。
下面的方案描述了圆形井的te殊情况,但是在凹槽的情况下分布相似

附加信息:
盖玻片的尺寸可以按需修改
相同类型的凝胶可适应培养皿或多孔板
可以根据需要制造其他形状的三角形孔,方形孔或矩形孔
其他深度可根据要求制造
二、美国flexercell 基底刚度可调控的细胞拉伸加载培养系统
可拉伸细胞基底硬度控制培养皿(CellSoft™ 100mm Round Dishes)
Cells sense soft! CellSoft™ offers softer substrates to match the material properties of tissue niches to better meet the needs of biological laboratories wanting to grow their cells on native stiffness。
●弹性模量范围1-80kPa
直径100mm培养皿,总生长表面积为57cm2
●可选多孔培养板、60mm和100mm培养皿
●BioFlex® CellSoft标准6孔板
●在柔性基底上牵拉细胞
●腔室载玻片CellSoft
●表面蛋白包被,wu菌单包装
CellSoft培养板有很多不同的种类,如不同的硬度,不同的孔板,用于显微观察的腔室载玻片(圆形多孔板),共价包被CollagenI或其他蛋白,可对细胞进行静态或动态牵拉应力刺激。更重要的一点,新型的CellSoft培养板可以反复胰酶消化和再接种细胞,蛋白包被的表面可以重复使用多达三次。
●柔软的硅胶弹性体涂层培养皿。
●模量范围:1—80 kPa
●传代细胞系扩增的里想选择
●光学透明,可通过倒置或直立显微镜直接观察细胞(膜厚度:1000um)
●共价键合的表面:有氨基,胶原蛋白(I型或IV型),弹性蛋白,ProNectin(RGD)和层粘连蛋白(YIGSR)等包被涂层和未经处理的。
●低自体荧光,可用于免疫组织化学分析或荧光探针。
●室温下避光中或wu直射光下储存长达1年。
1、CellSoft®bioflex可牵张拉伸刚度可调整 (1-80kpa)培养板 —cellsoft bioflex 6 well cuture plates

·CellSoft® BioFlex® 6-well or 24 well flexible culture plates to stretch cells on soft substrates
·Moduli range: 0.1, 5, 10, 20, 40, 200 kPa (Can be customized as required)


2、CellSoft®静态刚度可调整 (1-80kpa)培养皿和多孔板
CellSoft® substrates are “softer” compared to uncoated glass or polystyrene culture plates.



背景:
“细胞喜欢柔软!” flexercell®创始人兼总裁Albert Banes博士说 ”这些年来,细胞培养发生了变化,但是,热门的两项创新是在柔性的生长表面上生长和拉伸细胞以及控制基材刚性的能力。 flexercell新的CellSoft™培养产品进一步推动了这些创新,我们知道研究人员会喜欢这款产品。”
“Cells like soft!” says flexercell® Founder and President, Dr. Albert Banes, Ph.D. “Cell culture has met with change over the years, but two of the hottest innovations have been the ability to grow and stretch cells on flexible growth surfaces and to control the rigidity of the substrate. Our new CellSoft™ culture ware advances those innovations even further, and we know researchers will appreciate this product.”
美国flexercell公司于1988年shou次商业化了弹性体生长表面,并提供了根据心脏,血管,肺或其他机械活性组织的力学来可控地拉伸细胞的方法装置设备。 与玻璃或聚苯乙烯培养板相比,这些弹性体表面“柔软”且可拉伸。 借助CellSoft™,flexercell®可以创建更柔软的基质,以匹配组织niche(干细胞微环境)的材料特性,并更好地满足希望在天然刚度基材上生长细胞的生物实验室的需求。
注释:干细胞周围的细胞形成像摇篮样的环境保护着干细胞,这一环境被称为niche。niche不仅给干细胞提供养分,同时还指导干细胞的行动,决定干细胞的分化方向。
flexercell® was first to commercialize elastomer growth surfaces in 1988 and to provide the means to controllably stretch cells according to the mechanics of the heart, blood vessels, lungs, or other mechanically active tissues. These elastomer surfaces are “soft” and stretchable compared to glass or polystyrene culture plates. With CellSoft™, flexercell® has created even softer substrates to match the material properties of tissue niches and better meet the needs of biological laboratories wanting to grow their cells on native stiffness substrates.
CellSoft™具有各种刚度和板格式(圆盘和多孔板)。 它可以与胶原I或其他基质共价键合,并已预先灭菌并可以使用。 CellSoft™100 mm圆形培养皿非常适合用于传代细胞和其他平板形式,包括柔性底部Bioflex®平板,以进行静态或动态拉伸型实验。
CellSoft培养板有很多不同的种类,如不同的硬度(弹性模量范围1-80kPa),可用于显微观察的腔室载玻片(圆形多孔板),共价包被Collagen I或其他蛋白,可对细胞进行静态或动态牵拉应力刺激。更重要的一点,新型的CellSoft™ 培养板可以反复胰酶消化和再接种细胞,蛋白包被的表面可以重复使用多达三次
●弹性模量范围1-80kPa
●可选多孔板、60mm和100mm培养板
●BioFlex® CellSoft标准6孔板
●在柔性基底上牵拉细胞
●腔室载玻片CellSoft
●表面蛋白包被,wu菌单包装
Amino,
Collagen (Type I or IV),
Elastin,
ProNectin (RGD),
and Laminin (YIGSR)
and untreated (未处理)
CellSoft培养板有很多不同的种类,如不同的硬度,不同的孔板,用于显微观察的腔室载玻片(圆形多孔板),共价包被CollagenI或其他蛋白,可对细胞进行静态或动态牵拉应力刺激。更重要的一点,新型的CellSoft培养板可以反复胰酶消化和再接种细胞,蛋白包被的表面可以重复使用多达三次。

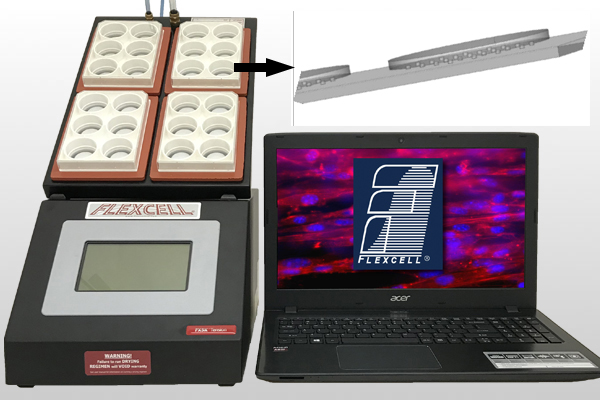
与flexercell FX-5000或6000T细胞牵张拉伸系统结合,可实现基底硬度控制牵张拉伸。

亮点:
1)该系统对二维、三维细胞和组织各种培养物提供轴向和圆周应力加载;不但具有双轴向拉伸力加载,还具备单轴向加力功能
2)计算机控制的应力加载系统,为体外培育的细胞提供j确的、可控制的、可重复的、静态的或者周期性的应力变化。
3)使用真空泵,抻拉培养板底部的弹性硅胶模,细胞培养板底部高伸展度可达到33%,通过气体装置可以自动调节和控制应力。
4)基于柔性膜基底变形、受力均匀;
5)可实时观察细胞、组织在应力作用下的反应;
6)具的flexstop隔离阀可使同一块培养板力的一部分培养孔的细胞受力,一部分培养孔的细胞不受力,方便对比实验;
7)与压力传导仪整合,同时兼备多通道细胞压力加载功能;
8)与Flex Flow平行板流室配套,可在牵拉细胞的同时施加流体切应力;
9)多达4通道,可4个不同程序同时运行,进行多个不同拉伸形变率对比实验;
10)同一程序中可以运行多种频率,多种振幅和多种波形;
11)加载模拟波形种类丰富:静态波形、正旋波形、心动波形、三角波形、矩形以及各种te制波形;
12)更好地控制在超低或超高应力下的波形;
13)电脑系统对牵张拉伸力加载周期、大小、频率、持续时间j确智能调控
14)加载分析各种细胞在牵张拉应力刺激下的生物化学反应
15)伸展度范围广:0-33%
16)牵拉频率范围广:0.01-5Hz
17)成功文献达4000多篇,国内150家单位使用。
三、细胞微图案牵张拉伸、流体剪切应力加载培养系统
美国flexercell基底拓扑微图案牵张拉伸培养系统,拓扑微图案压缩和流体剪切应力,细胞微图案牵张系统,细胞微图案压缩系统,细胞微图案流体剪切应力系统
型号:美国flexercell基底拓扑微图案
品牌:美国flexercell
美国flexercell基底拓扑微图案牵张拉伸培养系统
美国flexercell微图案压缩和流体剪切应力系统
细胞的生命活动受到胞外信号分子的调控,这些信号分子主要包括生物化学信号(激素、维生素等)及物理信号(弹性、流体剪切力、基质拓扑结构等),探究细胞是如何感知胞外信号分子并如何做出反应是细胞生物学研究的重点之一。细胞胞外拓扑结构是对细胞生存微环境形貌学的总称,生物体体内的许多组织都含有天然的...
(另提供flexercell基底拓扑微图案压缩和流体剪切应力培养系统)
美国新推出细胞微图案柔性基底膜牵张拉伸培养板 (6孔和24孔),使牵张拉伸培养板具有仿生表面形貌,细胞能根据纳米图案的方向上延伸生长。
纳米图案化牵张、压缩培养表面提供细胞微环境,模仿天然细胞外基质的对齐结构,促进细胞结构和功能发展。
-
仿生对齐的纳米级表面形貌
-
纳米形貌取向
亮点:微图案可按需求定制
亮点:
1)该系统对二维、三维细胞和组织各种培养物提供轴向和圆周应力加载;不但具有双轴向拉伸力加载,还具备单轴向加力功能
2)计算机控制的应力加载系统,为体外培育的细胞提供j确的、可控制的、可重复的、静态的或者周期性的应力变化。
3)使用真空泵,抻拉培养板底部的弹性硅胶模,细胞培养板底部高伸展度可达到33%,通过气体装置可以自动调节和控制应力。
4)基于柔性膜基底变形、受力均匀;
5)可实时观察细胞、组织在应力作用下的反应;
6)具的flexstop隔离阀可使同一块培养板力的一部分培养孔的细胞受力,一部分培养孔的细胞不受力,方便对比实验;
7)与压力传导仪整合,同时兼备多通道细胞压力加载功能;
8)与Flex Flow平行板流室配套,可在牵拉细胞的同时施加流体切应力;
9)多达4通道,可4个不同程序同时运行,进行多个不同拉伸形变率对比实验;
10)同一程序中可以运行多种频率,多种振幅和多种波形;
11)加载模拟波形种类丰富:静态波形、正旋波形、心动波形、三角波形、矩形以及各种te制波形;
12)更好地控制在超低或超高应力下的波形;
13)电脑系统对牵张拉伸力加载周期、大小、频率、持续时间j确智能调控
flexercell拓扑微图案压缩系统
美国新推出细胞微图案柔性基底膜压缩培养板 (6孔),使压缩培养板具有仿生表面形貌,细胞能根据纳米图案的方向上延伸生长。
纳米图案化牵张、压缩培养表面提供细胞微环境,模仿天然细胞外基质的对齐结构,促进细胞结构和功能发展。
-
仿生对齐的纳米级表面形貌
-
纳米形貌取向
亮点
1)该系统对各种组织、三维细胞培养物提供周期性或静态的压力加载;
2)基于柔性膜基底变形、受力均匀;
3)可实时观察细胞、组织在压力作用下的反应;
4)可有选择性地封阻对细胞的应力加载;
8)更好地控制在超低或超高应力下的波形;
9)多种波形种类:静态波形、正旋波形、心动波形、三角波形、矩形以及各种te制波形;
10)电脑系统对压力加载周期、大小、频率、持续时间j确智能调控
flexercell拓扑微图案流体剪切应力系统
四、单细胞限制器-cell confiner(Cell confinement)
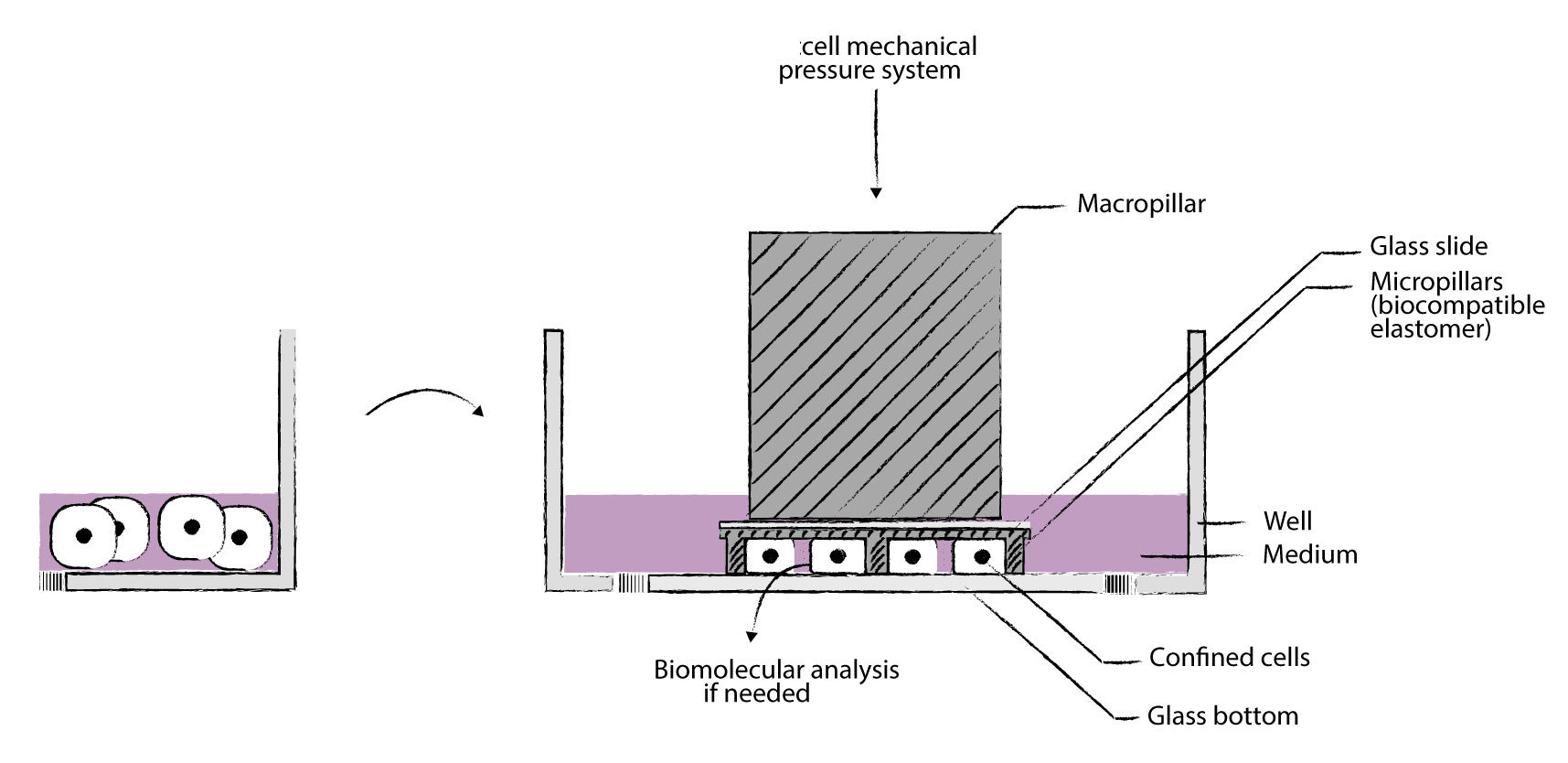
我们的cell Confiner是一种多功能设备,可通过对细胞应用定义明确的约束条件来研究细胞力学。限制方法基于将细胞固定在两个平行表面之间,从而实现均匀且定义明确的物理参数,例如细胞几何形状和环境弹性。此外,可以使用高分辨率显微镜对受限的细胞进行成像,因为该设备是光学透明的,并将细胞保持在焦平面上。
细胞被均匀地限制/压缩在两个亚微米分辨率的两个平行表面之间。两个表面之间的空间由微型PDMS支柱控制。 微型支柱在载玻片上制造,载玻片连接到PDMS活塞(吸盘)上。 活塞由真空泵控制,因此限制区的高度也受到控制。不同的限制高度(例如1um – 300um),允许长期细胞培养和细胞增殖,同时保持对封闭的完美控制与高分辨率光学显微镜系统兼容,可以处理足够多的细胞以进行完整的基因表达分析,可与生物功能化的微结构化底物和/或不同的基质(几何形状控制)结合使用
可以与凝胶结合(硬度控制),兼容任何细胞培养底物(培养皿至96孔板)。
产品特性:
>定义细胞的厚度和形状
用正确的限制滑片控制细胞的厚度
>同时进行多个实验
能够研究不同的细胞或同时应用不同的限制条件
>适用于高分辨率显微镜
光学透明的材料和紧凑的设计可实现高分辨率显微镜
>控制限制速度通过真空泵j确控制限制速度
>可逆限制:限制后取回您的细胞
由于细胞的非破坏性方法,可以进行分子分析
>与您自己的实验兼容
该限制器是一种小型设备,直接放置在您的细胞培养液顶部
1、典型应用:单细胞机械压缩刺激系统图
典型应用:
>癌症浸润测定:迁移行为和迁移转变的量化
>癌症侵袭性测定:体细胞或癌细胞的收缩力定量
>内吞作用测定:更好地观察膜发生的事件
>胞吐法测定:更好地观察在顶端膜发生的事件
>吞噬功能失调:机制的表征
>孔中的免疫系统:非粘附免疫细胞的二维迁移和相互作用
>免疫细胞相互作用:非贴壁免疫细胞的2D相互作用
>有丝分裂组装测定:有丝分裂纺锤体疾病的定量
>定量细胞迁移测定:细胞迁移特性的快速,精细分析
>癌症研究转移细胞的迁移
转移中的细胞收缩
DNA DSB修复(机械诱导)
基因组不稳定(细胞分裂)
分离共培养
>免疫学
免疫细胞迁移
非粘附细胞的成像
>器官生理学
癌细胞迁移
具有硬度控制的细胞区分
伤口愈合
分离共培养
细胞压缩反应
>罕见疾病
细胞核完整性
>老化
细胞核完整性
自噬相关疾病
>观测化
非粘附细胞的成像
细胞器的平面成像
>基础研究
细胞体积(细胞周期)
细胞机械力刺激反应
二维心肌细胞成熟测定
二维肝小管化验
3D心肌细胞成熟测定
3D肝小管测定
附着球体测定
细胞收缩力测定
细胞迁移测定
细胞核挤压测定
细胞j化
细胞体积测量
趋化性测定
共培养测定
胞吞试验
胞吐法
外泌体测定
片状脂蛋白和丝状体含量测定
活细胞成像
巨噬细胞j化测定
MT依赖性运输测定
神经肌肉连接测定
井中的神经元网络
细胞器定位分析
初次纤毛测定
骨骼肌细胞测定
平滑肌细胞
伤口愈合测定
-
Confinement and Low Adhesion Induce Fast Amoeboid Migration of Slow Mesenchymal Cells
Y.-J. Liu, M. Piel, Cell, et al., 2015 160(4), 659-672 -
Actin flows induce a universal coupling between cell speed and cell persistence
P. Maiuri, R. Voituriez, et al., Cell, 2015 161(2), 374–386 -
Geometric friction directs cell migration
M. Le Berre, M. Piel, et al., Physical Review Letter 2013 111, 198101 -
Mitotic rounding alters cell geometry to ensure efficient spindle assembly
O. M. Lancaster, B. Baum, et al., Developmental Cell, 2013 25(3), 270-283 -
Fine Control of Nuclear Confinement Identifies a Threshold Deformation leading to Lamina Rupture and Induction of Specific Genes
M. Le Berre, J. Aubertin, M. Piel, Integrative Biology, 2012 4 (11), 1406-1414 -
Exploring the Function of Cell Shape and Size during Mitosis
C. Cadart, H. K. Matthews, et al., Developmental Cell, 2014 29(2), 159-169 -
Methods for Two-Dimensional Cell Confinement
M. Le Berre, M. Piel, et al., 2014, Micropatterning in Cell Biology Part C, Methods in cell biology, 121, 213-29
References
[1] D. Huh, G. A. Hamilton, and D. E. Ingber, “From 3D cell culture to organs-on-chips,” Trends Cell Biol., vol. 21, no. 12, pp. 745–754, 2011.
[2] M. Ravi, V. Paramesh, S. R. Kaviya, E. Anuradha, and F. D. Paul Solomon, “3D cell culture systems: Advantages and applications,” J. Cell. Physiol., vol. 230, no. 1, pp. 16–26, 2015.
[3] J. W. Haycock, 3D cell culture: a review of current approaches and techniques., vol. 695. 2011.
[4] F. Pampaloni, E. G. Reynaud, and E. H. K. Stelzer, “The third dimension bridges the gap between cell culture and live tissue.,” Nat. Rev. Mol. Cell Biol., vol. 8, no. 10, pp. 839–845, 2007.
[5] J. Lee, M. J. Cuddihy, and N. A. Kotov, “Three-dimensional cell culture matrices: state of the art.,” Tissue Eng Part B Rev, vol. 14, no. 1, pp. 61–86, 2008.
[6] M. Vinci et al., “Advances in establishment and analysis of three-dimensional tumor spheroid-based functional assays for target validation and drug evaluation,” BMC Biol., vol. 10, no. 1, p. 29, 2012.
[7] B. A. Justice, N. A. Badr, and R. A. Felder, “3D cell culture opens new dimensions in cell-based assays,” Drug Discov. Today, vol. 14, no. 1–2, pp. 102–107, 2009.
[8] I. Meyvantsson and D. J. Beebe, “Cell culture models in microfluidic systems.,” Annu. Rev. Anal. Chem., vol. 1, pp. 423–449, 2008.
[9] E. W. K. Young and D. J. Beebe, “Fundamentals of microfluidic cell culture in controlled microenvironments,” Chem Soc Rev, vol. 39, no. 3, pp. 1036–1048, 2010.
[10] D. J. Beebe, G. a Mensing, and G. M. Walker, “Physics and applications of microfluidics in biology.,” Annu. Rev. Biomed. Eng., vol. 4, pp. 261–286, 2002.
[11] J. El-Ali, P. K. Sorger, and K. F. Jensen, “Cells on chips.,” Nature, vol. 442, no. 7101, pp. 403–411, 2006.
[12] J. Guck et al., “Optical deformability as an inherent cell marker for testing malignant transformation and metastatic competence,” Biophys J, vol. 88, no. 5, pp. 3689–3698, 2005.
[13] S. K?ster et al., “Drop-based microfluidic devices for encapsulation of single cells.,” Lab Chip, vol. 8, no. 7, pp. 1110–1115, 2008.
[14] H. Andersson and A. Van den Berg, “Microfluidic devices for cellomics: A review,” Sensors Actuators, B Chem., vol. 92, no. 3, pp. 315–325, 2003.
[15] M. W. Tibbitt and K. S. Anseth, “Hydrogels as extracellular matrix mimics for 3D cell culture,” Biotechnol. Bioeng., vol. 103, no. 4, pp. 655–663, 2009.
[16] J. P. Vacanti and R. Langer, “Tissue engineering: the design and fabrication of living replacement devices for surgical reconstruction and transplantation.,” Lancet, vol. 354, p. SI32-I34, 1999.
[17] G. S. D. Hetal Patel, Minal Bonde, “Biodegradable polymer scaffolds for tissue engineering,” Trends Biomater. Artif. Organs, vol. 25, no. 1, pp. 20–29, 2011.
[18] L. G. Griffith and M. A. Swartz, “Capturing complex 3D tissue physiology in vitro.,” Nat. Rev. Mol. cell Biol., vol. 7, no. 3, pp. 211–24, 2006.
[19] D. J. Tobin, “Scaffolds for Tissue Engineering and 3D Cell Culture,” Methods Mol. Biol., vol. 695, no. 2, pp. 213–227, 2011.
[20] J. Naranda et al., “Polyester type polyHIPE scaffolds with an interconnected porous structure for cartilage regeneration,” Sci. Rep., vol. 6, no. February, p. 28695, 2016.
[21] B. Dhandayuthapani, Y. Yoshida, T. Maekawa, and D. S. Kumar, “Polymeric scaffolds in tissue engineering application: A review,” Int. J. Polym. Sci., vol. 2011, no. ii, 2011.
[22] F. J. O’Brien, “Biomaterials & scaffolds for tissue engineering,” Mater. Today, vol. 14, no. 3, pp. 88–95, 2011.
[23] A. L. Paguirigan and D. J. Beebe, “Microfluidics meet cell biology: Bridging the gap by validation and application of microscale techniques for cell biological assays,” BioEssays, vol. 30, no. 9, pp. 811–821, Sep. 2008.
[24] F.-Q. Nie, M. Yamada, J. Kobayashi, M. Yamato, A. Kikuchi, and T. Okano, “On-chip cell migration assay using microfluidic channels.,” Biomaterials, vol. 28, no. 27, pp. 4017–4022, 2007.
[25] A. Valster, N. L. Tran, M. Nakada, M. E. Berens, A. Y. Chan, and M. Symons, “Cell migration and invasion assays,” Methods, vol. 37, no. 2, pp. 208–215, 2005.
[26] C. R. Justus, N. Leffler, M. Ruiz-Echevarria, and L. V Yang, “In vitro cell migration and invasion assays.,” J. Vis. Exp., vol. 752, no. 88, p. e51046, 2014.
[27] N. Kramer et al., “In vitro cell migration and invasion assays.,” Mutat Res, vol. 752, no. 1, pp. 10–24, 2013.
[28] J. W. Hong, V. Studer, G. Hang, W. F. Anderson, and S. R. Quake, “A nanoliter-scale nucleic acid processor with parallel architecture.,” Nat. Biotechnol., vol. 22, no. 4, pp. 435–439, 2004.
[29] J. Q. Boedicker, L. Li, T. R. Kline, and R. F. Ismagilov, “Detecting bacteria and determining their susceptibility to antibiotics by stochastic confinement in nanoliter droplets using plug-based microfluidics.,” Lab Chip, vol. 8, no. 8, pp. 1265–1272, 2008.
[30] G. Velve-Casquillas, M. Le Berre, M. Piel, and P. T. Tran, “Microfluidic tools for cell biological research,” Nano Today, vol. 5, no. 1. pp. 28–47, 2010.
[31] C. R. Terenna et al., “Physical Mechanisms Redirecting Cell Polarity and Cell Shape in Fission Yeast,” Curr. Biol., vol. 18, no. 22, pp. 1748–1753, Nov. 2008.
[32] G. Faure-andré, “Regulation of Dendritic Cell Migration by CD74, the MHC Class II–Associated Invariant Chain,” Science (80-. )., vol. 1705, no. December, 2008.
[33] S. M. McFaul, B. K. Lin, and H. Ma, “Cell separation based on size and deformability using microfluidic funnel ratchets,” Lab Chip, vol. 12, no. 13, pp. 2369–2376, 2012.
[34] S. C. Hur, N. K. Henderson-MacLennan, E. R. B. McCabe, and D. Di Carlo, “Deformability-based cell classification and enrichment using inertial microfluidics.,” Lab Chip, vol. 11, no. 5, pp. 912–920, 2011.
[35] H. W. Hou, Q. S. Li, G. Y. H. Lee, A. P. Kumar, C. N. Ong, and C. T. Lim, “Deformability study of breast cancer cells using microfluidics,” Biomed. Microdevices, vol. 11, no. 3, pp. 557–564, 2009.
