高保真基因文库制备仪
高通量单细胞、单分子、纳米颗粒乳化液滴封装与分选系统
wei依可实现单分子、细胞、细胞器、纳米颗粒封装和高吞吐量分选。 DNA封装使得能够以前所未有的速度筛选大型复杂的基因库。
制备型

是一种多功能且用户友好的仪器,用于制备活的哺乳动物或微生物细胞、细胞器、DNA 或其他生物材料以进行高分辨率下游分析。
使用我们的 试剂盒, 安quan快速地将活细胞、细胞器或 DNA 片段连同测定化学物质一起封装在高度稳定的皮升体积双乳化液滴中。这些液滴通过移液、涡旋、孵育、流式细胞术、分选,甚至长期储存都很稳定。更重要的是,可以从液滴中回收细胞进行扩增。
与适当的分析技术一起, 应用包括:
? 人体免疫细胞、NK细胞
? 微生物细胞的酶分泌
? 哺乳动物细胞分泌细胞因子
? 验证基因编辑,包括评估意外的在靶和脱靶重排
借助 ,研究人员可以更轻松地深入了解人类、动物、植物和微生物的细胞功能和基因组学。 还可以为其他工作流程生成单乳液液滴,包括封装 DNA 以进行wu偏差的quan基因组扩增。 我们提供 所需的quan套试剂、试剂盒和附件。此外,用户培训是我们标准 安装服务的一部分。
you势
? 可以将活的哺乳动物或微生物细胞包裹在高度稳定的双乳液滴中,用于功能测定、孵育、表型分析和筛选。
? 帮助 回答工程细胞和基因治疗研究中的关键问题。
? 支持分子工程工作流程,包括酶进化和途径工程。
? 具有用户友好的界面和较小的占地面积,使每个分子生物学实验室都能使用微流体的强大功能。
双层乳化试剂盒
试剂都包含在试剂盒内,每次操作能封装数百万个细胞或分子。

Sort制备分选型

这个 家族的新成员可以在用户友好的工作流程中生成和分选双乳液液滴,不需要任何基于荧光的细胞分选仪的经验。
工作流程从高度稳定的液滴中捕获 100 kb DNA 片段、小单细胞或其他生物材料开始,用于 PCR 和酶活性评估等测定。然后,根据液滴内包含物荧光,对液滴进行分选,以仅获取感兴趣的样品以进行高分辨率下游分析,例如:
验证没有隐藏意外重排的 PCR 偏差的 CRISPR 编辑
识别病毒和转基因整合位点
在单细胞水平评估酶活性
执行高通量 GFP 筛选
you势:
· 能够有针对性地富集长片段
· 高灵敏度 - 单分子检测
· 保持DNA的内在性
· 几乎不需要先验序列知识
· 兼容PacBio、Nanopore和Illumina测序
· 通过自动微流体系统轻松设置
· 将周转时间从数周缩短到数天
应用:
· 结构变化
· 复杂样本(例如癌症活检)
· 难以测序的区域(例如富含GC)
· 基因组间隙闭合
· 基因定相/基因分型/单倍体分型
· 将抗性基因与宿主联系起来
· 低丰度区域的测序
· 重复区域的评估
· 仅含有纳克 DNA 的样品
辅助材料
1、DE50 墨盒-用于将哺乳动物细胞包裹在双乳液滴中

产生高度稳定的 ~100 皮升双乳液滴,以封装哺乳动物细胞,将大细胞转化为单细胞检测。使用 DE50 Cartridge 的工作流程包括细胞因子分泌和细胞杀伤测定。只需加载试剂和样品,将试剂盒放入 ,然后运行程序。
同时或单du运行 8 个样品
在 8 分钟内每条泳道产生多达 500,000 个液滴在 CO 2培养箱中用细胞孵育液滴
恢复选定的单元格进行扩展
2、DE20 墨盒-用于将微生物细胞、细胞器或 DNA 长片段包裹在双乳液滴中
使用du立的 DE20 小柱产生高度稳定的约 1.5 皮升的双乳化液滴,以封装数百万个微生物细胞、细胞器或 DNA。每个液滴充当单细胞分辨率测定或 DNA 靶向富集的隔室。只需加载试剂和样品,将试剂盒放入 或 Sort,然后运行程序。
同时或单du运行 8 个样品
在 45 分钟内每条泳道产生多达 1000 万个液滴在 CO 2培养箱中用细胞孵育液滴
恢复选定的单元格进行扩展确保wu污染的工作流程
3、DE20 分选盒-用于分选含有 DNA 或小细胞的双乳液滴

使用新型 DE20 分选盒分选含有荧光 DNA 或小细胞的双乳化液滴。这个用户友好的系统专为与我们的新仪器 Sort 一起使用而设计,不需要荧光分选仪方面的专业知识。只需加载试剂和样品,将试剂盒放入 Sort,然后运行分选程序。
同时运行多达 8 个样品每天分拣数十亿个液滴确保wu污染的工作流程
4、SE85 墨盒-用于将 DNA、RNA、细胞器或小细胞包裹在单乳液液滴中
使用du立的 SE85 墨盒产生单乳液液滴以封装数百万个分子、细胞器或细胞。每个液滴充当单分子或单细胞分辨率测定的隔室。只需加载试剂和样品,将试剂盒放入 或 Sort,然后运行程序。
同时或单du运行 8 个样品
在 40 秒内每条泳道产生超过 50,000 个液滴确保wu污染的工作流程
应用
使用的专有微流体技术来生成高度稳定的双乳化液和单乳化液滴,用于封装生物材料。
这种多功能且用户友好的仪器封装了生命的组成部分,例如活的哺乳动物和微生物细胞,或 DNA 长片段,用于一系列下游目的,包括孵化、分析、分类和分子谱分析。
Sort 以 专有的微流体技术为基础,增加了基于封装生物材料的荧光分选双乳液滴的能力。
以下是一些zui具代表性的应用。
细胞功能分析:
· 人体免疫细胞、NK细胞
· 微生物细胞的酶分泌
· 哺乳动物细胞分泌细胞因子
基因组学应用:
· 结构变化
· 复杂样本(例如癌症活检)
· 难以测序的区域(例如富含GC)
· 基因组间隙闭合
· 基因定相/基因分型/单倍体分型
· 将抗性基因与宿主联系起来
· 低丰度区域的测序
· 重复区域的评估
· 仅含有纳克 DNA 的样品
植物基因组学应用:
· 丰富了植物品种中的特定基因组区域
· 揭示没有参考基因组的大麦品种中生物合成基因簇的结构
· 添补番茄品种中生物合成基因簇结构的空白
应用实例:
1、quan基因组的准确扩增
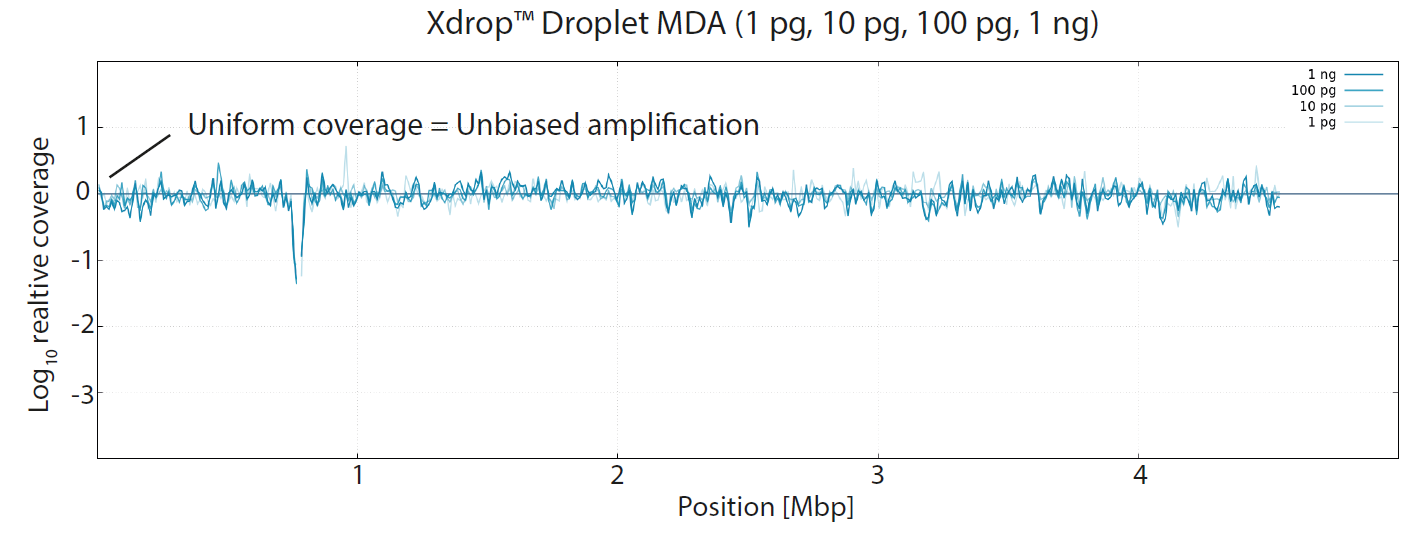
事实上,99%的靶基因组不止一次被文库的测序读取所覆盖。
使用 ? dMDA 技术扩增基因,即使在输入量低至 1 pg 时也能保真准确的扩增quan步基因。
2、基因定相Phasing
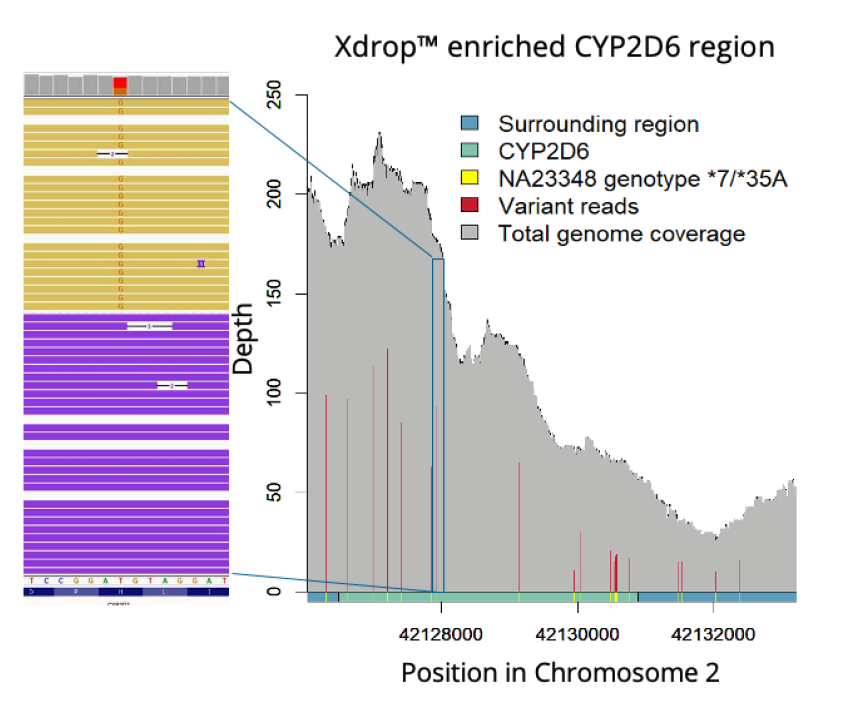
CYP2D6*7等位基因包含1个单核苷酸多态性(SNP),而CYP2D6*25A包含22个SNP。
技术能够准确测出 23 种变体,包括Phasing(见下图)。
?通过简单的设计和长DNA片段(~100 kb)的间接序列捕获为应用提供支持。结合长读长测序,?克服了在单个遗传异质样品中基因定相的困难。
富集的高保真度能够检测等位基因中的一个或多个SNP,并使从头组装能够解决结构变化。
3、监测基因复杂的结构变化
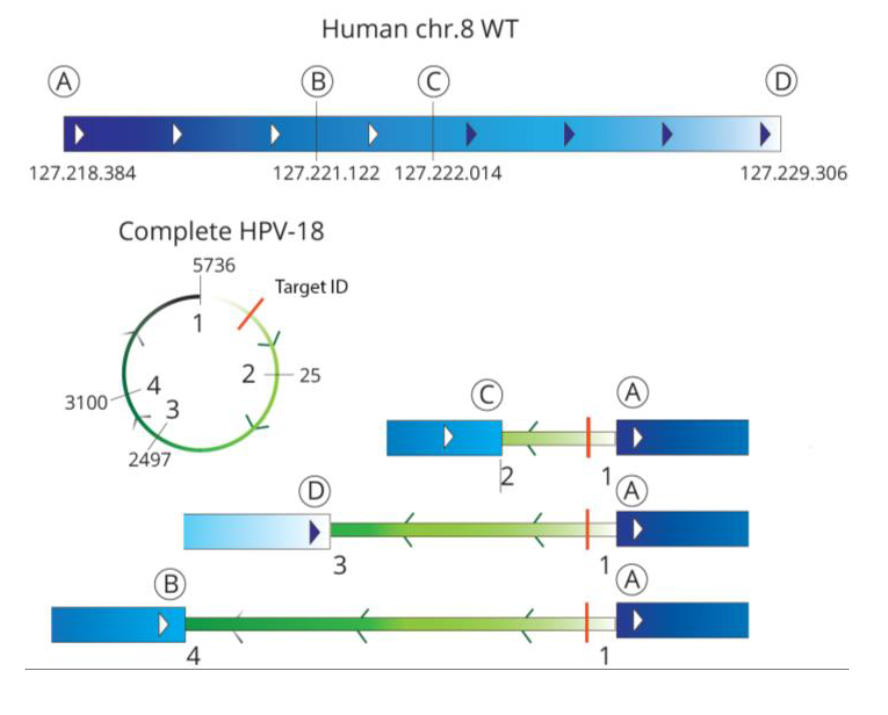
对跨越断点的读数的详细分析显示,在8号染色体上的区域中存在多个HPV18整合位点。PacBio、Oxford Nanopore和Illumina数据集支持已识别的整合位点。
?技术是一种新的靶向DNA富集方法,其du特之处在于可以选择、富集和测序天然DNA片段。这允许富集大的基因组部分(约100kb),包括未知区域。使用?方法生成的长片段不仅适用于长读测序,还可以为短读测序提供有价值的上下文信息。
4、对未知的感兴趣区域(ROI)的测序
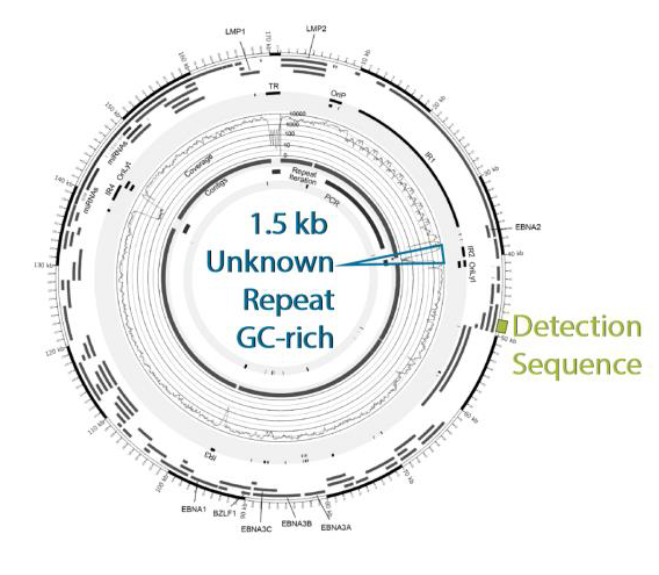
PCR和基于探针的靶向DNA富集方法需要密集的设计you化和完整的感兴趣区域(ROI)知识。这些传统方法在表征复杂的基因组环境(例如重复区域、结构变异、富含GC的区域)以及未知和重排的区域方面也失败。为了克服这些限制,开发了一种新的微流体方法,即基于间接序列捕获的?富集工作流。
?可以富集需要在检测序列上设计单个引物对的长(~100 kb)靶DNA区域,对应于ROI的一小部分或侧翼区域。该扩增子专门用于检测、选择和富集quan长ROI,zui终进行捕获和测序。
使用?技术解决这些挑战性区域的能力为解决许多其他挑战性区域打开了大门,这些区域可能代表了很大比例的基因组。将?富集与长读和短读测序技术相结合,可以提供所需的高分辨率,以快速且经济的方式解决复杂的基因组场景,绕过quan基因组测序。
5、识别和确认CRISPR基因编辑操作

通过简单的设计和长DNA片段的间接序列捕获,有助于识别通常难以找到且成本高昂的转基因插入位点。
构建体中的随机整合和宿主基因组序列的存在使整合位点鉴定变得复杂。 使验证基因组工程变得简单明了。
实验过程
1、混合 dPCR 试剂

将DNA添加到含有目标特异性pri-mers的dPCR反应混合物中。
2、片上液滴生产

反应在液滴发生器芯片上分配,以生产水包水液(DE)液滴。
3、高分辨率微滴式聚合酶链反应
液滴经过热循环,可并行运行数百万个PCR-re操作。通过检测荧光液滴,可以识别携带目标DNA分子的液滴。
4、计数和分离液滴
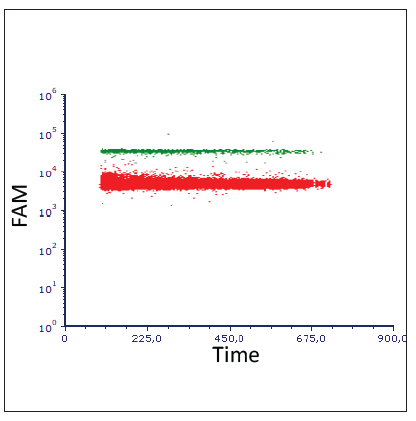
检测和分离使用细胞分选仪(FACS)完成,该分选仪可以收集含有靶DNA分子的PCR阳性液滴
5、富集DNA的扩增

长DNA片段的富集单分子通过液滴多位移扩增(dMDA)进行扩增,以确保wu偏的DNA扩增和对目标区域的均匀覆盖。
参考文献
1. Population-wide gene disruption in the murine lung epithelium via AAV-mediated delivery of CRISPR-Cas9 components Honglin Chen, Steffen Durinck, Hetal Patel, etc.
Mol. Ther. Methods Clin. Dev. 2022. 27: 431–449; doi: 10.1016/j.omtm.2022.10.016.
2. Target-enriched nanopore sequencing and de novo assembly reveals co-occurrences of complex on-target genomic rearrangements induced by CRISPR-Cas9 in human cells Keyi Geng, Lara G Merino, Linda Wedemann, etc.
Genome Res. 2022. 32(10): 1876–1891; doi: 10.1101/gr.276901.122
3. Characterization of FMR1 repeat expansion and intragenic variants by indirect sequence capture Valentina Grosso, Luca Marcolungo, Simone Maestri,etc.
Front. Genet. 2021. 12: 743230; doi: 10.3389/fgene.2021.743230
4. Generation and analysis of innovative genomically humanized knockin SOD1, TARDBP (TDP-43), and FUS mouse models Anny Devoy, Georgia Price, Francesca De Giorgio,etc.
iScience. 2021. 24(12): 103463; doi: 10.1016/j.isci.2021.103463
5. CRISPR/Cas9 deletions induce adverse on-target genomic effects leading to functional DNA in human cells Keyi Geng, Lara Garcia Merino, Linda Wedemann, etc.
bioRxiv 2021.07.01.450727; doi: https://doi.org/10.1101/2021.07.01.450727
6. Alt-RPL36 downregulates the PI3K-AKT-mTOR signaling pathway by interacting with TMEM24 Xiongwen Cao, Alexandra Khitun, Yang Luo, etc.
Nature Communications 12, 508, 2021. doi: 10.1038/s41467-020-20841-6
7. Reconstruction of the birth of a male sex chromosome present in Atlantic herring Rafati N, Chen J, Herpin A, etc.
Proc Natl Acad Sci U S A. 2020 Sep 29;117(39):24359-24368. doi: 10.1073/pnas.2009925117. Epub 2020 Sep 16. PMID: 32938798.
8. Verification of CRISPR edng and finding transgenic inserts by Indirect sequence capture followed by short- and long- read sequencing Blondal Thorarinn, Gamba Cristina, Jagd Lea M?ller, etc.
Methods. 2021 Jul;191:68-77. doi: 10.1016/j.ymeth.2021.02.003. Epub 2021 Feb 12. PMID: 33582298.
9. : Targeted sequencing of long DNA molecules from low input samples using droplet sorting Madsen EB, H?ijer I, Kvist T, etc.
2020 Sep;41(9):1671-1679. doi: 10.1002/humu.24063. Epub 2020 Jun 29. PMID: 32516842; PMCID: PMC7496172.
10. Cell-based Long-read whole genome analysis of human single cells Joanna H?rd, Jeff E Mold, Jesper Eisfeldt,etc.
2021bioRxiv 2021.04.13.439527; doi: https://doi.org/10.1101/2021.04.13.439527
11. Corrigendum to "Generation of a set of isogenic, gene-edited iPSC lines homozygous for all main APOE variants and an APOE knock-out line Schmid B, Prehn KR, Nimsanor N, etc.
Stem Cell Res. 2020 Sep 21;48:102005. doi: 10.1016/j.scr.2020.102005. Epub ahead of print. Erratum for: Stem Cell Res. 2019 Jan;34:101349. PMID: 32971461.